- PCR is een methode die wordt gebruikt om DNA uit een kleine hoeveelheid DNA-sjabloon te amplificeren.RT-PCR maakt gebruik van omgekeerde transcriptie om een DNA-sjabloon te produceren uit een RNA-bron die vervolgens kan worden geamplificeerd.
- PCR en RT-PCR zijn doorgaans eindpuntreacties, terwijl qPCR en RT-qPCR de kinetiek van de snelheid van productsynthese tijdens de PCR-reactie gebruiken om de hoeveelheid aanwezige template te kwantificeren.
- Nieuwere methoden, zoals digitale PCR, bieden absolute kwantificering van de initiële DNA-sjabloon, terwijl methoden zoals isothermische PCR de behoefte aan dure apparatuur verminderen om betrouwbare resultaten te verkrijgen.
Polymerasekettingreactie (PCR) is een relatief eenvoudige en veelgebruikte techniek in de moleculaire biologie om DNA- en RNA-sequenties te amplificeren en te detecteren.Vergeleken met traditionele methoden voor het klonen en amplificeren van DNA, die vaak dagen kunnen duren, heeft PCR slechts een paar uur nodig.PCR is zeer gevoelig en vereist een minimale template voor detectie en amplificatie van specifieke sequenties.Fundamentele PCR-methoden zijn verder gevorderd dan eenvoudige DNA- en RNA-detectie.Hieronder geven we een overzicht van de verschillende PCR-methoden en de reagentia die we bij Enzo Life Sciences ter beschikking stellen voor uw onderzoeksbehoeften.We streven ernaar wetenschappers te helpen snel toegang te krijgen tot PCR-reagentia voor gebruik in hun volgende onderzoeksproject!
PCR
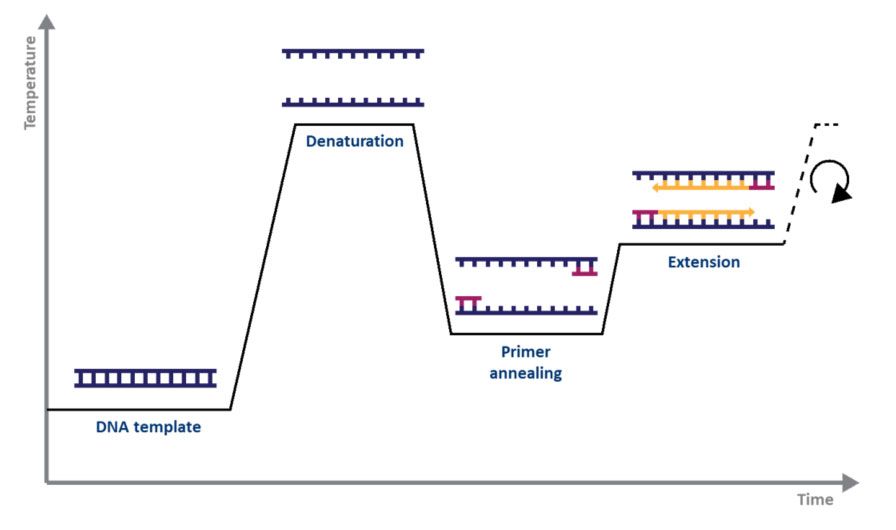
Voor standaard PCR heeft u alleen een DNA-polymerase, magnesium, nucleotiden, primers, de te amplificeren DNA-sjabloon en een thermocycler nodig.Het PCR-mechanisme is net zo eenvoudig als het doel ervan: 1) dubbelstrengig DNA (dsDNA) wordt door hitte gedenatureerd, 2) primers worden uitgelijnd met de enkele DNA-strengen, en 3) de primers worden verlengd door DNA-polymerase, wat resulteert in twee kopieën van de DNA-strengen. originele DNA-streng.Het denaturatie-, uitgloei- en verlengingsproces over een reeks temperaturen en tijden staat bekend als één amplificatiecyclus (Fig. 1).
| Figuur 1.Schematische weergave van een amplificatiecyclus door PCR. |
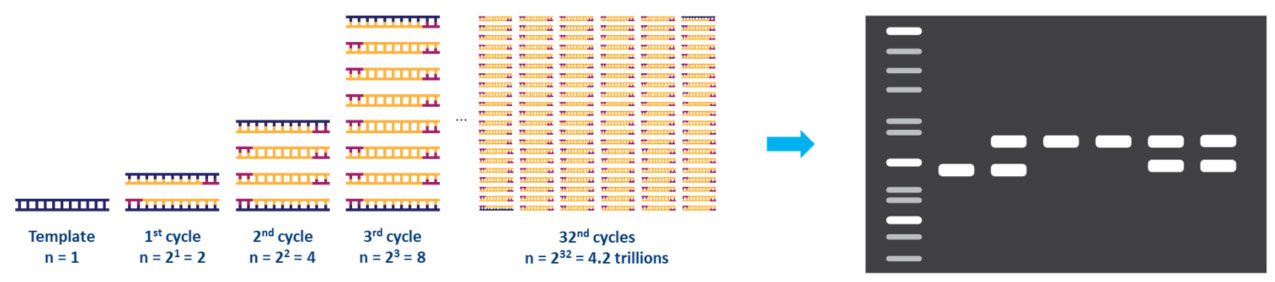
Elke stap van de cyclus moet worden geoptimaliseerd voor de gebruikte template en primerset.Deze cyclus wordt ongeveer 20-40 keer herhaald en het geamplificeerde product kan vervolgens worden geanalyseerd, doorgaans met agarosegel (Fig. 2).
| Figuur 2.Amplificatie van een DNA-matrijs door PCR en analyse door agarosegelelektroforese. |
Omdat PCR een zeer gevoelige methode is en er voor afzonderlijke reacties zeer kleine volumes nodig zijn, wordt de bereiding van een mastermix voor meerdere reacties aanbevolen.Het mastermengsel moet goed worden gemengd en vervolgens worden gesplitst op basis van het aantal reacties, waarbij ervoor moet worden gezorgd dat elke reactie dezelfde hoeveelheid enzym, dNTP's en primers bevat.Veel leveranciers, zoals Enzo Life Sciences, bieden ook PCR-mixen aan die alles al bevatten behalve primers en de DNA-template.
Guanine/cytosinerijke (GC-rijke) regio's vormen een uitdaging bij standaard PCR-technieken.GC-rijke sequenties zijn stabieler dan sequenties met een lager GC-gehalte.Bovendien hebben GC-rijke sequenties de neiging secundaire structuren te vormen, zoals haarspeldlussen.Als gevolg hiervan zijn GC-rijke dubbele strengen moeilijk volledig te scheiden tijdens de denaturatiefase.Bijgevolg kan DNA-polymerase de nieuwe streng niet ongehinderd synthetiseren.Een hogere denaturatietemperatuur kan dit verbeteren, en aanpassingen in de richting van een hogere uitgloeitemperatuur en een kortere uitgloeitijd kunnen de aspecifieke binding van GC-rijke primers voorkomen.Aanvullende reagentia kunnen de amplificatie van GC-rijke sequenties verbeteren.DMSO, glycerol en betaïne helpen de secundaire structuren te verstoren die worden veroorzaakt door GC-interacties en vergemakkelijken daardoor de scheiding van de dubbele strengen.
Hotstart-PCR
Niet-specifieke amplificatie is een probleem dat kan optreden tijdens PCR.De meeste DNA-polymerasen die voor PCR worden gebruikt, werken het beste bij temperaturen rond de 68°C tot 72°C.Het enzym kan echter ook bij lagere temperaturen actief zijn, zij het in mindere mate.Bij temperaturen ver onder de annealingstemperatuur kunnen primers niet-specifiek binden en tot niet-specifieke amplificatie leiden, zelfs als de reactie op ijs wordt uitgevoerd.Dit kan worden voorkomen door polymeraseremmers te gebruiken die pas dissociëren van het DNA-polymerase zodra een bepaalde temperatuur is bereikt, vandaar de term hot start PCR.De remmer kan een antilichaam zijn dat het polymerase bindt en denatureert bij de initiële denaturatietemperatuur (typisch 95°C).
High-fidelity-polymerase
Hoewel DNA-polymerasen redelijk nauwkeurig amplificeren tot de oorspronkelijke templatesequentie, kunnen er fouten optreden bij het matchen van nucleotiden.Mismatches in toepassingen zoals klonen kunnen resulteren in ingekorte transcripten en verkeerd vertaalde of inactieve eiwitten stroomafwaarts.Om deze mismatches te voorkomen, zijn polymerasen met een “proeflees”-activiteit geïdentificeerd en in de workflow opgenomen.Het eerste proefleespolymerase, Pfu, werd in 1991 geïdentificeerd in Pyrococcus furiosus.Dit Pfu-enzym heeft een 3'-naar-5'-exonuclease-activiteit.Terwijl het DNA wordt geamplificeerd, verwijdert het exonuclease niet-overeenkomende nucleotiden aan het 3'-uiteinde van de streng.Het juiste nucleotide wordt dan vervangen en de DNA-synthese gaat door.De identificatie van onjuiste nucleotidesequenties is gebaseerd op de bindingsaffiniteit voor het juiste nucleosidetrifosfaat met het enzym, waarbij inefficiënte binding de synthese vertraagt en de juiste vervanging mogelijk maakt.De proefleesactiviteit van Pfu-polymerase resulteert in minder fouten in de uiteindelijke sequentie vergeleken met Taq DNA-polymerase.In de afgelopen jaren zijn andere proefleesenzymen geïdentificeerd en zijn er wijzigingen aan het oorspronkelijke Pfu-enzym aangebracht om het foutenpercentage tijdens DNA-amplificatie verder te verminderen.
RT-PCR
Omgekeerde transcriptie-PCR, of RT-PCR, maakt het gebruik van RNA als sjabloon mogelijk.Een extra stap maakt de detectie en amplificatie van RNA mogelijk.Het RNA wordt omgekeerd getranscribeerd naar complementair DNA (cDNA), met behulp van reverse transcriptase.De kwaliteit en zuiverheid van de RNA-template zijn essentieel voor het succes van RT-PCR.De eerste stap van RT-PCR is de synthese van een DNA/RNA-hybride.Reverse transcriptase heeft ook een RNase H-functie, die het RNA-gedeelte van de hybride afbreekt.Het enkelstrengige DNA-molecuul wordt vervolgens gecompleteerd door de DNA-afhankelijke DNA-polymerase-activiteit van het reverse transcriptase tot cDNA.De efficiëntie van de eerstestrengsreactie kan het amplificatieproces beïnvloeden.Vanaf nu wordt de standaard PCR-procedure gebruikt om het cDNA te amplificeren.De mogelijkheid om RNA via RT-PCR om te zetten in cDNA heeft veel voordelen en wordt voornamelijk gebruikt voor analyse van genexpressie.RNA is enkelstrengig en zeer onstabiel, wat het een uitdaging maakt om mee te werken.Het dient gewoonlijk als een eerste stap in qPCR, die RNA-transcripten in een biologisch monster kwantificeert.
qPCR en RT-qPCR
Kwantitatieve PCR (qPCR) wordt gebruikt voor het detecteren, karakteriseren en kwantificeren van nucleïnezuren voor tal van toepassingen.Bij RT-qPCR worden RNA-transcripten vaak gekwantificeerd door ze eerst omgekeerd in cDNA te transcriberen, zoals hierboven beschreven, en vervolgens wordt qPCR uitgevoerd.Net als bij standaard PCR wordt DNA geamplificeerd door drie zich herhalende stappen: denaturatie, annealing en verlenging.Bij qPCR maakt fluorescerende labeling echter het verzamelen van gegevens mogelijk naarmate de PCR vordert.Deze techniek heeft veel voordelen vanwege het scala aan beschikbare methoden en chemie.
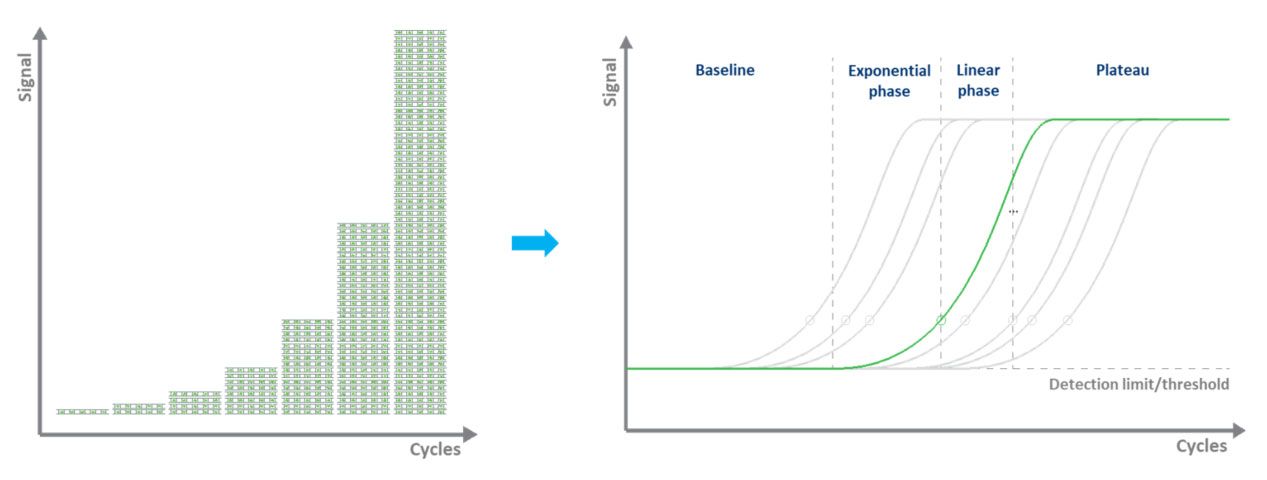
Bij op kleurstof gebaseerde qPCR (meestal groen) maakt fluorescerende labeling de kwantificering van de geamplificeerde DNA-moleculen mogelijk door gebruik te maken van een dsDNA-bindende kleurstof.Tijdens elke cyclus wordt de fluorescentie gemeten.Het fluorescentiesignaal neemt proportioneel toe met de hoeveelheid gerepliceerd DNA.Daarom wordt het DNA in “real-time” gekwantificeerd (Fig. 3).De nadelen van op kleurstof gebaseerde qPCR zijn dat er slechts één doelwit tegelijk kan worden onderzocht en dat de kleurstof zich zal binden aan eventueel ds-DNA dat in het monster aanwezig is.
| Figuur 3.Het amplificeren van een DNA-sjabloon door qPCR en het in realtime meten van het fluorescentiesignaal. |
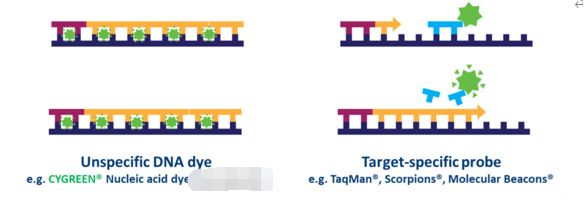
Bij op probes gebaseerde qPCR kunnen veel doelen tegelijkertijd in elk monster worden gedetecteerd, maar dit vereist optimalisatie en ontwerp van doelspecifieke probe(s) die naast primers worden gebruikt.Er zijn verschillende soorten sondeontwerpen beschikbaar, maar het meest voorkomende type is een hydrolysesonde, die een fluorofoor en quencher bevat.Fluorescentie-resonantie-energieoverdracht (FRET) voorkomt de emissie van de fluorofoor via de quencher terwijl de sonde intact is.Tijdens de PCR-reactie wordt de probe echter gehydrolyseerd tijdens primerverlenging en amplificatie van de specifieke sequentie waaraan deze is gebonden.De splitsing van de probe scheidt de fluorofoor van de quencher en resulteert in een amplificatie-afhankelijke toename in fluorescentie (Fig. 4).Het fluorescentiesignaal van een op probes gebaseerde qPCR-reactie is dus evenredig met de hoeveelheid van de probe-doelsequentie die in het monster aanwezig is.Omdat op probes gebaseerde qPCR specifieker is dan op kleurstoffen gebaseerde qPCR, is het vaak de technologie die wordt gebruikt in op qPCR gebaseerde diagnostische testen.
| Figuur 4.Verschillen tussen op kleurstoffen gebaseerde en op probes gebaseerde qPCR. |
Isotherme versterking
De hierboven genoemde PCR-technieken vereisen dure thermocyclingapparatuur om de kamertemperaturen nauwkeurig op en neer te laten gaan voor de denaturatie-, annealing- en verlengingsstappen.Er is een aantal technieken ontwikkeld waarvoor dergelijke nauwkeurige apparaten niet nodig zijn en die kunnen worden uitgevoerd in een eenvoudig waterbad of zelfs in de betreffende cellen.Deze technieken worden gezamenlijk isothermische versterking genoemd en werken op basis van exponentiële, lineaire of cascadeversterking.
Het bekendste type isothermische amplificatie is lusgemedieerde isotherme amplificatie, oftewel LAMP.LAMP maakt gebruik van exponentiële amplificatie bij 65⁰C om template-DNA of RNA te amplificeren.Bij het uitvoeren van LAMP worden vier tot zes primers gebruikt die complementair zijn aan gebieden van het doel-DNA met een DNA-polymerase om nieuw DNA te synthetiseren.Twee van deze primers hebben complementaire sequenties die sequenties in de andere primers herkennen en deze binden, waardoor een ‘lus’-structuur kan ontstaan in het nieuw gesynthetiseerde DNA dat vervolgens helpt bij het hybridiseren van de primer in daaropvolgende amplificatieronden.LAMP kan op meerdere manieren worden gevisualiseerd, waaronder fluorescentie, agarosegelelektroforese of colorimetrie.Het gemak van het visualiseren en detecteren van de aan- of afwezigheid van een product door middel van colorimetrie en het ontbreken van dure apparatuur die nodig was, maakten LAMP tot een geschikte optie voor SARS-CoV-2-testen in gebieden waar klinische laboratoriumtests niet direct beschikbaar waren, of voor opslag en transport van monsters niet haalbaar was, of in laboratoria die voorheen niet over PCR-thermocyclingapparatuur beschikten.
Posttijd: 19 augustus 2023