Uitgangsmateriaal: RNA
Kwantitatieve reverse transcriptie-PCR (RT-qPCR) is een experimentele methode die wordt gebruikt in PCR-experimenten waarbij RNA als uitgangsmateriaal wordt gebruikt.Bij deze methode wordt eerst totaal RNA of messenger RNA (mRNA) getranscribeerd in complementair DNA (cDNA) door middel van reverse transcriptase.Vervolgens werd een qPCR-reactie uitgevoerd met het cDNA als sjabloon.RT-qPCR is gebruikt in een verscheidenheid aan toepassingen in de moleculaire biologie, waaronder analyse van genexpressie, validatie van RNA-interferentie, validatie van microarrays, detectie van pathogenen, genetische tests en ziekteonderzoek.
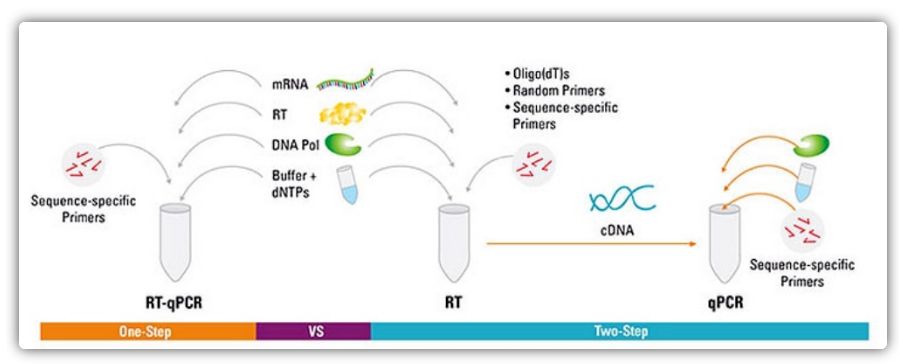
Eenstaps- en tweestapsmethoden voor RT-qPCR
RT-qPCR kan worden uitgevoerd met een eenstaps- of tweestapsmethode.Eenstaps RT-qPCR combineert reverse transcriptie en PCR-amplificatie, waardoor reverse transcriptase en DNA-polymerase de reactie in dezelfde buis onder dezelfde bufferomstandigheden kunnen voltooien.Eenstaps RT-qPCR vereist alleen het gebruik van sequentiespecifieke primers.In tweestaps RT-qPCR worden reverse transcriptie en PCR-amplificatie uitgevoerd in twee buizen, met behulp van verschillende geoptimaliseerde buffers, reactieomstandigheden en primerontwerpstrategieën.
| Voordeel | Nadeel | |
| Een stap | Deze methode heeft minder experimentele fouten omdat beide reacties in één buis worden gedaan
Minder pipetteerstappen verminderen het risico op verontreiniging
Geschikt voor high-throughput versterking/screening, snel en reproduceerbaar | Reacties in twee stappen kunnen niet afzonderlijk worden geoptimaliseerd
Aangezien de reactieomstandigheden worden aangetast door de tweestapsreactie te combineren, is de gevoeligheid niet zo goed als die van de tweestapsmethode
Het aantal doelen dat door een enkele steekproef wordt gedetecteerd, is klein |
| Twee stappen | Mogelijkheid om stabiele cDNA-bibliotheken te creëren die voor lange tijd kunnen worden bewaard en in meerdere reacties kunnen worden gebruikt
Doelgenen en referentiegenen kunnen uit dezelfde cDNA-bibliotheek worden geamplificeerd zonder dat meerdere cDNA-bibliotheken nodig zijn
Reactiebuffers en reactiecondities die optimalisatie van enkele reactieruns mogelijk maken
Flexibele selectie van triggervoorwaarden | Het gebruik van meerdere buisjes en meer pipetteerstappen verhoogt het risico op DNA-besmetting, en tijdrovend.
Vereist meer optimalisatie dan de eenstapsmethode |
Gerelateerde producten:
RT-qPCR Easyᵀᴹ (One Step)-SYBR Groen I
RT-qPCR Easyᵀᴹ (één stap)-Taqman
RT Easyᵀᴹ I Master Premix voor eerste streng CDNA-synthese
Realtime PCR Easyᵀᴹ-SYBR Green I Kit
Selectie van totaal RNA en mRNA
Bij het ontwerpen van een RT-qPCR-experiment is het belangrijk om te beslissen of totaal RNA of gezuiverd mRNA als sjabloon voor reverse transcriptie moet worden gebruikt.Hoewel mRNA mogelijk een iets hogere gevoeligheid kan bieden, wordt totaal-RNA nog steeds vaak gebruikt.De reden hiervoor is dat totaal RNA als uitgangsmateriaal een belangrijker voordeel heeft dan mRNA.Ten eerste vereist het proces minder zuiveringsstappen, wat zorgt voor een beter kwantitatief herstel van de sjabloon en een betere normalisatie van de resultaten voor het starten van celaantallen.Ten tweede vermijdt het de mRNA-verrijkingsstap, die de mogelijkheid van scheve resultaten als gevolg van verschillende terugwinningen van verschillende mRNA's kan voorkomen.Over het algemeen is, aangezien in de meeste toepassingen de relatieve kwantificatie van het doelgen belangrijker is dan de absolute gevoeligheid van de detectie, in de meeste gevallen totaal RNA geschikter.
Omgekeerde transcriptieprimer
Bij de tweestapsmethode kunnen drie verschillende methoden worden gebruikt om de cDNA-reactie te primen: oligo(dT)-primers, willekeurige primers of sequentiespecifieke primers.Gewoonlijk worden oligo(dT)-primers en willekeurige primers in combinatie gebruikt.Deze primers versmelten met de template-mRNA-streng en bieden reverse transcriptase een startpunt voor synthese.
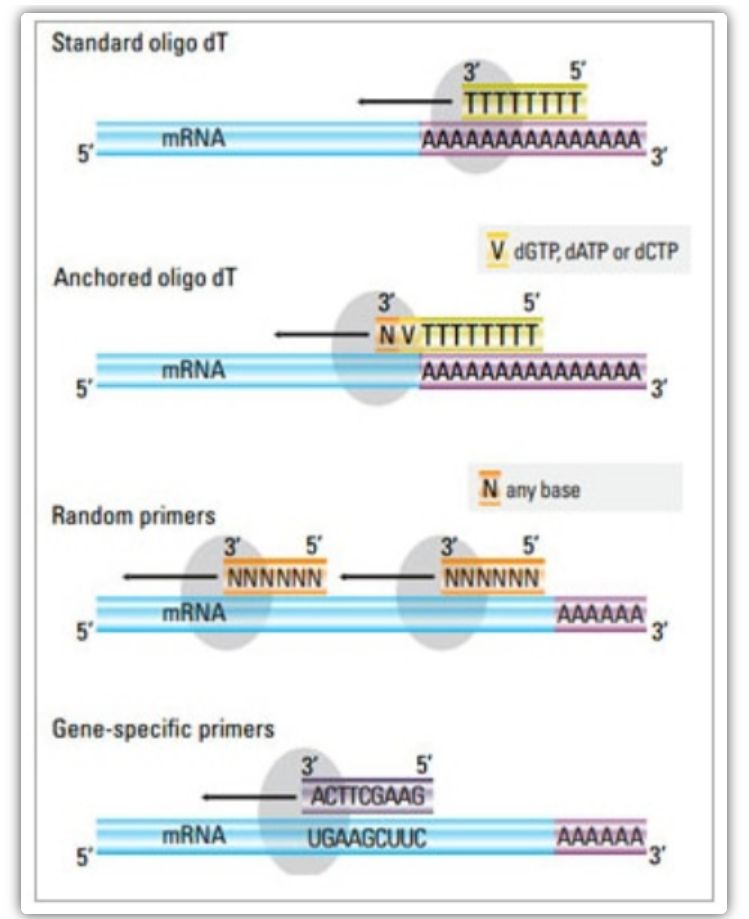
| Primer selectie | Structuur en functie | Voordeel | Nadeel |
| Oligo(dT) primer (of verankerde oligo(dT) primer) | Uitgebreide versmelting met thymineresiduen aan de poly(A)-staart van mRNA;anker oligo (dT) primer bevat een G, C of A aan het 3'-uiteinde (ankerplaats) | Synthese van cDNA van volledige lengte uit mRNA met poly(A)-staart
Van toepassing wanneer er minder uitgangsmateriaal beschikbaar is
Verankeringsplaats zorgt ervoor dat de oligo(dT)-primer bindt aan de 5'-poly(A)-staart van het mRNA | Alleen geschikt voor het amplificeren van genen met poly(A)-staarten
Verkrijg cDNA dat is afgekapt vanaf de priming-site*2 in poly(A)
Vooringenomen om te binden aan het 3'-uiteinde *
*Deze mogelijkheid wordt geminimaliseerd als verankerde oligo(dT)-primers worden gebruikt |
| willekeurige primer
| 6 tot 9 basen lang, die tijdens RNA-transcriptie aan meerdere sites kunnen hechten | Anneal aan alle RNA's (tRNA, rRNA en mRNA)
Geschikt voor transcripties met een significante secundaire structuur, of wanneer er minder uitgangsmateriaal beschikbaar is
Hoge cDNA-opbrengst | cDNA wordt omgekeerd getranscribeerd van al het RNA, wat meestal niet gewenst is en het signaal van het doel-mRNA kan verdunnen
krijg afgeknot cDNA |
| sequentiespecifieke primers | Aangepaste primers gericht op specifieke mRNA-sequenties | specifieke cDNA-bibliotheek
Verbeter de gevoeligheid
Omgekeerde qPCR-primers gebruiken | Alleen beperkt tot de synthese van een enkel doelgen |
Omgekeerde transcriptase
Reverse transcriptase is een enzym dat RNA gebruikt om DNA te synthetiseren.Sommige reverse transcriptasen hebben RNase-activiteit en kunnen na transcriptie RNA-strengen in hybride RNA-DNA-strengen afbreken.Als het geen RNase-enzymatische activiteit heeft, kan RNaseH worden toegevoegd voor een hogere qPCR-efficiëntie.Veelgebruikte enzymen zijn onder meer Moloney murine leukemievirus reverse transcriptase en aviair myeloblastoomvirus reverse transcriptase.Voor RT-qPCR is het ideaal om een reverse transcriptase te kiezen met een hogere thermostabiliteit, zodat cDNA-synthese kan worden uitgevoerd bij hogere temperaturen, waardoor een succesvolle transcriptie van RNA's met een hogere secundaire structuur wordt gegarandeerd, terwijl hun volledige activiteit behouden blijft tijdens de reactie, wat resulteert in hogere cDNA-opbrengsten.
Gerelateerde producten:
Foreasy M-MLV omgekeerde transcriptase
RNase H-activiteit van reverse transcriptase
RNaseH kan RNA-strengen afbreken van RNA-DNA-duplexen, waardoor een efficiënte synthese van dubbelstrengs DNA mogelijk wordt.Wanneer echter lang mRNA als matrijs wordt gebruikt, kan het RNA voortijdig worden afgebroken, wat resulteert in afgeknot cDNA.Daarom is het vaak gunstig om RNaseH-activiteit tijdens cDNA-klonering te minimaliseren als synthese van lange transcripten gewenst is.Daarentegen zijn reverse transcriptasen met RNase H-activiteit vaak gunstig voor qPCR-toepassingen omdat ze het smelten van RNA-DNA-duplexen tijdens de eerste cyclus van PCR verbeteren.
Primer-ontwerp
PCR-primers die worden gebruikt voor de qPCR-stap in RT-qPCR zouden idealiter ontworpen moeten zijn om een exon-exon-overgang te overspannen, waar een amplificatieprimer mogelijk een daadwerkelijke exon-intron-grens zou kunnen overspannen.Aangezien intron-bevattende genomische DNA-sequenties niet worden geamplificeerd, verkleint dit ontwerp het risico van geamplificeerde valse positieven door besmettend genomisch DNA.
Als primers niet kunnen worden ontworpen om exons of exon-exon-grenzen te scheiden, kan het nodig zijn om RNA-monsters te behandelen met RNase-vrij DNase I of dsDNase om genomische DNA-verontreiniging te verwijderen.
RT-qPCR-controle
Een reverse transcription negatieve controle (-RT-controle) moet worden opgenomen in alle RT-qPCR-experimenten om DNA-verontreiniging te detecteren (zoals genomisch DNA of PCR-producten van eerdere reacties).Deze controle bevat alle reactiecomponenten behalve reverse transcriptase.Aangezien reverse transcriptie niet optreedt bij deze controle, is verontreiniging door DNA het meest waarschijnlijk als PCR-amplificatie wordt waargenomen.
Posttijd: 02-08-2022








